Images
Científicos del IRB Barcelona desarrollan una estrategia computacional para proteínas potenciales regulados por la quinasa Aurora A.
La quinasa Aurora A, un oncogén, está involucrada en la regulación de los aspectos clave del ciclo celular. Sin embargo, se sabe muy poco de cómo esta proteína realiza sus muchas funciones celulares y las proteínas a las que regula, etc. Un equipo del IRB Barcelona, liderado por el investigador ICREA Patrick Aloy ha desarrollado , en colaboración con el Centro de Regulación Genómica, una estrategia computacional para identificar nuevas moléculas regularas por fosforilación por Aurora A. El trabajo se ha publicado en EMBO reports, una prestigiosa revista del Nature Publishing Group.
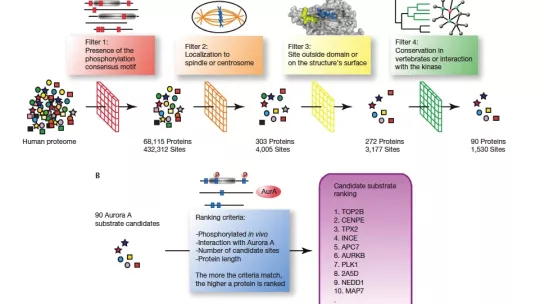
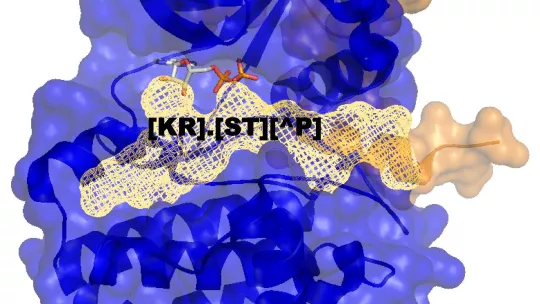
Aurora A pertenece a un grupo de proteínas que se encargan de regular la actividad de otras, controlando así las funciones más básicas de las células. “Solo se conocen 17 proteínas a las que modula y estas no pueden explicar todas las funciones que se le conocen”, explica Patrick Aloy. Por ello, estos investigadores abordaron la cuestión diseñando una estrategia computacional para buscar posibles dianas de esta proteína. La herramienta consiste en hacer pasar las moléculas por “filtros” virtuales, que seleccionan a las que presentan características que les permitirían interaccionar y ser fosforiladas por Aurora A. Encontraron 90 candidatos posibles y de estos escogieron 10 al azar para comprobar sus predicciones en sistemas biológicos. El resultado fue que 8 de estas moléculas realmente interaccionaban con la proteína de estudio, lo que significa que su método de predicción tiene una fiabilidad del 80%.
Conocer bien las distintas funciones de esta proteína tiene una gran importancia ya que se ha visto que Aurora A está sobre-expresada en algunos tipos de cáncer. Además, en caso de que exista el cáncer, estos niveles elevados están asociados a un mal pronóstico de la enfermedad. Hace tiempo que se trabaja en la búsqueda de fármacos que bloqueen la acción de Aurora A y de hecho ya hay variaos compuestos terapéuticos que se está probando en ensayos clínicos en humanos. La identificación de sustratos de Aurora A presentados es este estudio, contribuirán a contextualizar sus funciones y a entender mejor los posibles efectos secundarios causados por su inhibición.
Artículo de referencia:
Uncovering new substrates for Aurora A kinase.
Teresa Sardon, Roland A Pache, Amelie Stein, Henrik Molin, Isabelle Vernos & Patrick Aloy.
EMBO Reports (2010, 11(12): 977-984) [DOI: 10.1038/embor.2010.17]
IRB Barcelona
El Instituto de Investigación Biomédica (IRB Barcelona) trabaja para conseguir una vida libre de enfermedades. Desarrolla una investigación multidisciplinar de excelencia para curar el cáncer y otras enfermedades vinculadas al envejecimiento. Establece colaboraciones con la industria farmacéutica y los principales hospitales para hacer llegar los resultados de la investigación a la sociedad, a través de la transferencia de tecnología, y realiza diferentes iniciativas de divulgación científica para mantener un diálogo abierto con la ciudadanía. El IRB Barcelona es un centro internacional que acoge alrededor de 400 científicos de más de 30 nacionalidades. Reconocido como Centro de Excelencia Severo Ochoa desde 2011, es un centro CERCA y miembro del Barcelona Institute of Science and Technology (BIST).