Images
E. coli és l’organisme model millor conegut a nivell molecular, s’usa intensivament per a la producció biotecnològica i és d’interès biomèdic per les soques patològiques infeccioses.
L’article de Nature Biotecnology és una col·laboració entre biòlegs, bioquímics i bioinformàtics de l’Institut Craig Venter, la Universitat de Virginia i l’Institut de Recerca Biomèdica (IRB).
L’equip de 18 investigadors presenta el primer plànol de les interaccions clau entre les peces moleculars del bacteri que permet començar a entendre com funciona la seva maquinària vital més íntima.
Investigadors de l’Institut J. Craig Venter i de la Virginia Commonwealth University dels Estats Units, en estreta col·laboració amb científics de l’Institut de Recerca Biomèdica (IRB Barcelona) publiquen el primer mapa de les interaccions moleculars entre proteïnes –anomenat interactoma- d’Escherichia coli (E. coli) que permet començar a comprendre com funciona, com treballa íntimament, el bacteri.
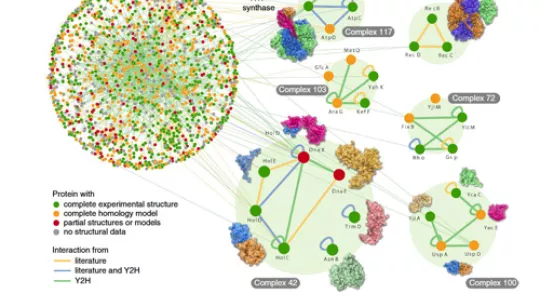
L’estudi revela al voltant del 25% (2.234) de les aproximadament 10.000 interaccions clau estimades que es donen a E. coli, i cobreix aproximadament el 70% del seu proteoma (la col.lecció de proteïnes).
L’estudi es publica a Nature Biotechnology, una de les revistes científiques de referència per a la comunitat científica, i precisament aquí perquè E. coli és el bacteri més usat en biotecnologia blanca, és a dir, per produir grans quantitats de material químic, com ara l’artemisina per tractar la malària o la insulina per a la diabetis.
D’E. coli se’n coneix extensivament la genòmica - hi ha desenes de soques seqüenciades, des de d’algunes patològiques a altres que no ho són-, se’n coneix la proteòmica de complexes (mapa dels complexes de proteïnes) i és l’organisme model on s’han fet més estudis de metabolòmica (el mapa dels cicles metabòlics).
La informació obtinguda d’aquests estudis –omics a gran escala ha permès obtenir el llistat dels elements moleculars d’E. coli que intervenen en el seu funcionament. Però per tenir el plànol total de l’ésser viu, de com treballa i funciona, calia encarar l’interactoma, les interaccions moleculars entre tots els elements descrits, i en concret, entre proteïnes.
“I això hem fet”, diu el bioinformàtic Patrick Aloy. “El mapa que hem presentat ens permet començar a veure el bacteri íntimament i ens pot permetre dissenyar, per exemple, antibiòtics per trencar interaccions entre proteïnes i desballestar parts de la seva maquinària molecular, o simplement ens permet entendre com funciona en tota la seva complexitat”, explica l’investigador ICREA de l’IRB.
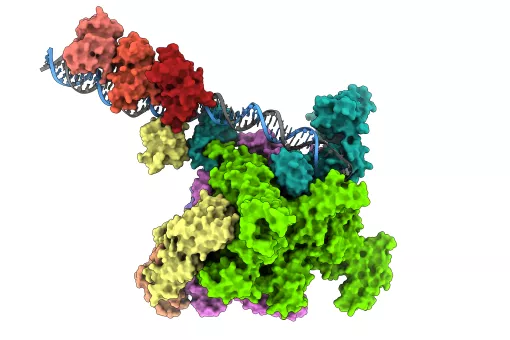
El seu grup en “Bioinformàtica estructural i biologia de xarxes” ha fet l’anàlisi computacional de les xarxes d’interaccions i n’han modelat les estructures tridimensionals. “Sense les estructures estàs cec i no pots intervenir. Aquest treball ens ajuda a comprendre com funcionen les xarxes que constitueixen i regulen les funcions vitals del bacteri”, diu Aloy.
El treball bioinformàtic és obra de Roberto Mosca i Arnaud Ceol, investigadors postdoctorals, del laboratori d’Aloy, amb dos metodologies generades al laboratori, Interactome3D i NetAligner. “El mapa del Roberto i l’Arnaud és al rovell de l’ou de l’article, ja que han permès l’anotació estructural i l’anàlisi bioinformàtic de les xarxes d’interacció, respectivament”, detalla Aloy.
L’última part de la feina ha estat confirmar “el que ja es sospitava”, explica Aloy, com ara que el bacteri E. coli és un bon organisme model i les conclusions són extrapolables a molts altres bacteris.
L’estudi inclou una comparativa amb els proteomes de 20 bacteris molts dels quals són el focus de patologies severes, com ara les produïdes per l’l’Helicobacter pylori, el Vibrio cholera, l’Haemophilus influenzae, el Neisseria meningitides i l’Yersinia pestis.
“Aportem informació útil per a usos biotecnològics, per entendre patologia i per dissenyar drogues”, conclou Aloy, qui manté diversos projectes amb l’Institut Craig Venter.
El disseny de nous fàrmacs per lluitar contra les infeccions era l’objectiu principal de l’AntiPathoGN, un projecte de la Unió Europea finalitzat el juny passat del qual en formava part el grup d’Aloy. Els resultats d’aquest article a Nature Biotechnology s’emmarquen també dins d’aquest projecte europeu, que en els propers mesos, encara generarà més resultats científics.
Article de referència:
The binary protein-protein interaction landscape of Escherichia coli
Seesandra V Rajagopala, Patricia Sikorski, Ashwani Kumar, Roberto Mosca, James Vlasblom, Roland Arnold, Jonathan Franca-Koh, Suman B Pakala, Sadhna Phanse, Arnaud Ceol, Roman Häuser, Gabriella Siszler, Stefan Wuchty, Andrew Emili, Mohan Babu, Patrick Aloy, Rembert Pieper & Peter Uetz.
Nature Biotechnology (2014) doi: 10.1038/nbt.2831
IRB Barcelona
L’Institut de Recerca Biomèdica (IRB Barcelona) treballa per aconseguir una vida lliure de malalties. Desenvolupa una recerca multidisciplinària d’excel·lència per curar el càncer i altres malalties vinculades a l'envelliment. Treballa establint col·laboracions amb la indústria farmacèutica i els principals hospitals per fer arribar els resultats de la recerca a la societat a través de la transferència de tecnologia, i du a terme diferents iniciatives de divulgació científica per mantenir un diàleg obert amb la ciutadania. L’IRB Barcelona és un centre internacional que acull al voltant de 400 investigadors de més de 30 nacionalitats. Reconegut com a Centre d'Excel·lència Severo Ochoa des de 2011, és un centre CERCA i membre del Barcelona Institute of Science and Technology (BIST).