Images
La visión que típicamente tenemos sobre las proteínas, la maquinaria molecular que lleva a cabo la mayoría de las funciones dentro de una célula, asume que poseen una estructura tridimensional bien definida. Los procesos celulares (por ejemplo la reproducción) son activados o desactivados de acuerdo a una coreografía molecular que implica la unión de unas proteínas a otras. En los últimos tiempos, se ha visto que incluso una proteína pequeña (digamos de unos 50 residuos) puede unirse a muchas otras proteínas – lo que resulta muy ventajoso para la célula, puesto que dependiendo de la identidad de la proteína con la que interactúe puede generar múltiples respuestas celulares. Paralelamente, cada vez resulta más evidente que una estructura estática es una representación muy pobre de una proteína, y que es más apropiado representarla como una colección de conformaciones (un ‘ensemble’, en inglés conjunto). Esto nos lleva a darnos cuenta de que la movilidad de las diferentes partes de las proteínas (la dinámica) es crítica para su funcionalidad. La cuestión que entonces se ha convertido en reto científico es cómo una proteína pequeña puede ser tan promiscua y cuáles son las bases moleculares que subyacen.
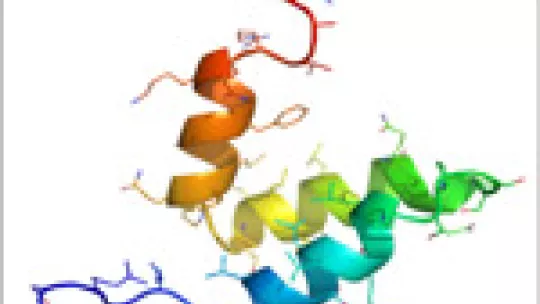
Para abordar este problema Athi N. Naganathan (becario Juan de la Cierva) y Modesto Orozco, del programa conjunto en Biología Computacional IRB Barcelona-BSC, eligieron como modelo el dominio de unión al DNA del coactivador nuclear (NCBD) de la proteína CREB (CBP, del inglés CREB binding protein), un pequeño dominio helicoidal de unos de 50 residuos. NCBD a pesar de su pequeño tamaño es un modelo único al mostrar una promiscuidad notable, conociéndose su unión a siete proteínas diferentes. Se han realizado extensas simulaciones de la dinámica molecular de este dominio, empleando tanto representaciones reducidas como modelos atomísticos complementados con restricciones obtenidas a partir de una amplia variedad de técnicas experimentales que aportan información sobre las dimensiones y la estructura del ‘ensemble’. Por primera vez han conseguido obtener una representación a nivel atómica sobre el ‘ensemble’ funcional de NCBD y, respondiendo a las cuestiones fundamentales sobre los mecanismos de unión de proteínas, han demostrado la coexistencia de múltiples conformaciones de las proteínas. Las simulaciones muestran que la transición de la estructura desplegada a la estructura completamente plegada de NCBD ocurre sin encontrar barreras de energía libre importantes. Han demostrado además cómo el plegamiento de NCBD está íntimamente ligado a sus requerimientos funcionales, lo que pone de relieve cómo un mecanismo de "reóstato molecular" permite conseguir multifuncionalidad en pequeños dominios de unión.
Reference articleThe Native Ensemble and Folding of a Protein Molten-Globule: Functional Consequence of Downhill Folding.
Athi N. Naganathan and Modesto Orozco.
JACS (dx.doi.org/10.1021/ja204053n)
IRB Barcelona
El Instituto de Investigación Biomédica (IRB Barcelona) trabaja para conseguir una vida libre de enfermedades. Desarrolla una investigación multidisciplinar de excelencia para curar el cáncer y otras enfermedades vinculadas al envejecimiento. Establece colaboraciones con la industria farmacéutica y los principales hospitales para hacer llegar los resultados de la investigación a la sociedad, a través de la transferencia de tecnología, y realiza diferentes iniciativas de divulgación científica para mantener un diálogo abierto con la ciudadanía. El IRB Barcelona es un centro internacional que acoge alrededor de 400 científicos de más de 30 nacionalidades. Reconocido como Centro de Excelencia Severo Ochoa desde 2011, es un centro CERCA y miembro del Barcelona Institute of Science and Technology (BIST).